TPPP 및 FAT1-LINC02374 유전자, eGFR 감소 연관성 확인돼 예후 마커로 활용
다중유전위험점수 구축, 고위험군 선별해 만성신장병 악화 예측 및 진단 가능해

특정 유전자의 변이가 만성신장병 악화에 영향을 미친다는 사실이 다기관 만성신장병 코호트 연구를 통해 세계 최초로 확인됐다.
최근 간단한 유전체 마커 검사를 통해 조기에 만성신장병의 악화를 예측·진단할 수 있는 길이 열렸다. 이를 활용해 고위험군을 선별해 집중적으로 치료하면 예후를 개선시킬 수 있다는 점에서 주목된다.
서울대병원 신장내과 오국환·서울의대 예방의학교실 박수경 교수 공동 연구팀이 다기관 만성신장병 코호트 환자의 임상 정보와 유전체 DB를 바탕으로 신장병의 악화 및 예후와 관련된 유전체 마커를 탐색하여 진단 시스템을 개발했다고 최근 밝혔다.
만성신장병(CKD, chronic kidney disease)은 3개월 이상 만성적으로 신장 기능이 지속적으로 저하되는 질병으로 전 세계 성인의 10% 이상이 영향을 받을 정도로 빠르게 증가하고 있다. 제때에 관리와 치료가 잘되지 않아 수년 동안에 걸쳐 신장 기능이 서서히 감소되면 투석 또는 신장이식을 받아야 하는 말기신부전에 이를 수 있다.
만성신장병의 신기능이 나빠지는 속도는 개인마다 큰 차이가 있지만 대개의 경우 5∼10년에 걸쳐 신장 기능이 악화된다. 때문에 만성신장병 초기에 진행 악화 및 투석 위험성이 큰 환자군을 조기에 예측하고 진단해 집중 관리할 수 있는 진단 시스템이 무엇보다 필요하다.
하지만 현재까지 정확한 진단 시스템이 부재했다. 만성신장병의 발병률이 유전적 요인과 관련이 있다고 알려져 있으나, 어떤 유전적 마커가 이 병의 진행에 영향을 미치는지는 정확하게 밝혀진 바가 없기 때문이다.
연구팀은 만성신장병 진행의 척도로 eGFR(추정 사구체여과율) 감소와 관련된 유전적 변이를 식별하기 위해 2011년 구축해 최장 10년간 추적을 지속하고 있는 한국의 다기관 만성신장병 코호트(KNOW-CKD)를 통해 만성신장병 환자 1738명을 대상으로 전장유전체 연관분석연구(GWAS)를 수행했다.
연구 결과, eGFR의 연간 변화를 나타내는 'eGFR 기울기'가 만성신장병 환자의 예후와 관련이 있는 것을 확인했다. 연구팀은 이 코호트 환자의 임상 정보와 유전체 DB를 바탕으로 신장병의 악화 및 예후인자인 eGFR 기울기와 관련된 유전체 마커를 탐색했다.
그 결과, 두 개의 새로운 유전자좌인 'TPPP 및 FAT1-LINC02374'에서 SNP(Single Nucleotide Polymorphism, 단일염기다형성)의 특정 변이 패턴이 신장병이 빠르게 악화하는 악화군에서 많이 발견되는 것을 세계 최초로 발견했다.
이는 연구팀이 발견한 'TPPP 및 FAT1-LINC02374' 유전자가 만성신장병 환자의 예후와 관련된 SNP 마커라는 것을 의미한다.
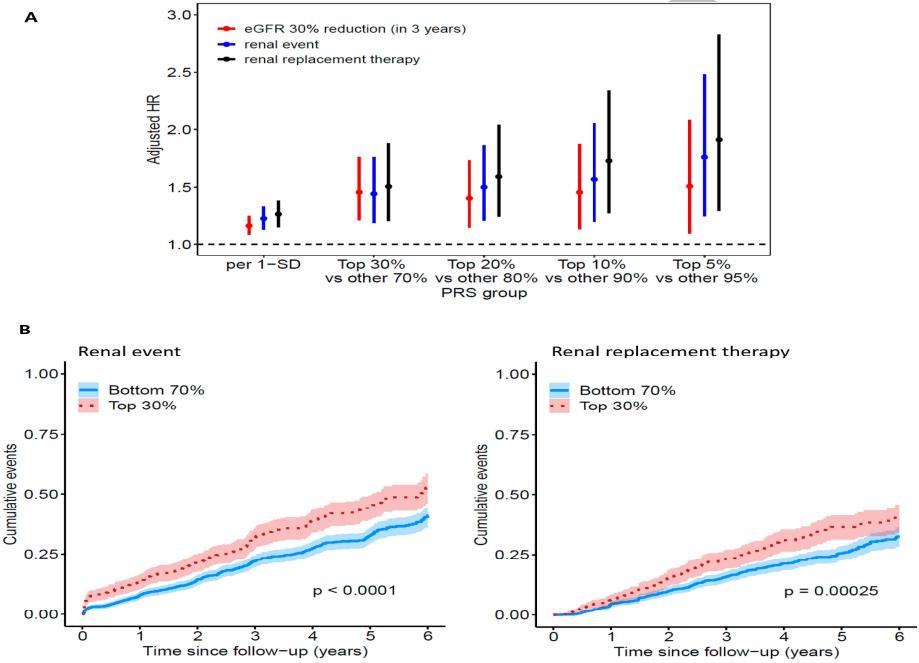
연구팀은 이 결과를 검증하기 위해 유럽인과 아프리카계로 구성된 만성신부전 코호트(CRIC) 연구에서 만성신장병 환자 2498명을 대상으로 eGFR 기울기와 관련된 P < 10-6으로 선택된 SNP에 대한 인종 간 재현성 연구(replication study)를 수행했다.
여러 발현 양적 특성 유전자좌(eQTL) 연구, 경로 농축 분석, 후성유전학적 구조의 탐색 및 전사인자 결합부위의 억제 예측을 통해 유전자좌의 잠재적인 생물학적 영향을 조사했다.
eQTL 연구는 재현성이 확인된 SNP가 신장 기능과 관련된 여러 유전자의 발현 수준을 조절한다는 것을 밝혔다. 이러한 SNP는 유전자 발현을 조절하는 인핸서 영역 근처에 위치해 전사인자의 결합을 방해한다. 이 결과는 TPPP 및 FAT1-LINC02374 유전자좌의 변이가 eGFR 기울기와 연관성이 있음을 시사한다.
연구팀은 이 중 독립적으로 유의한 상위 22개 SNP를 사용해 전사인자의 결합을 방해해 eGFR의 감소를 나타내는 다중유전위험점수(PRS)를 도출해냈다.
이를 바탕으로 연구팀은 만성신장병 환자의 신기능 급속 악화 및 말기신부전으로의 악화 등을 예측할 수 있는 다중유전위험점수(PRS)를 구축해 임상적 유용성을 확인했다고 설명했다.
오국환 교수는 "세계 최초로 발견한 이 TPPP 및 FAT1-LINC02374 유전자좌의 SNP 마커가 만성신장병 환자의 신장병 악화에 대한 예측 마커가 될 수 있음을 보여준다"며 "이를 기반으로 한 다중유전위험점수를 활용하면 만성신장병 환자 가운데 신기능이 빠르게 악화할 위험이 큰 환자군을 조기에 예측·선별이 가능해져 적시에 집중적인 관리와 치료가 가능해질 것으로 기대된다"고 말했다.
한편 이번 연구 결과는 신장학 분야 최고 권위 학술지인 <미국신장학회지(JASN, IF: 14.978)> 최신호에 게재됐다.


